Visualizzazione di set di dati chimici e analisi esplorativa dei dati [Python, RDKit]
Come passaggio preliminare alla creazione del modello di machine learning / QSAR, è importante analizzare il contenuto dei dati composti costituiti da un elenco di strutture e attività chimiche da una vista a volo d'uccello al fine di selezionare e comprendere il modello.
Questa volta, eseguiremo un'analisi esplorativa dei dati di set di dati composti come metodo di scienza dei dati per trovare le caratteristiche relative alla variabile obiettivo.
Cos'è l'analisi esplorativa dei dati?
L'analisi esplorativa dei dati è un approccio importante nelle prime fasi dell'analisi dei dati, che riassume le caratteristiche chiave attraverso la visualizzazione dei dati e l'analisi della correlazione.Viene eseguito per verificare se il modello originariamente previsto è riconosciuto nei dati o per cercare qualsiasi modello.
Il libro di Tukey dello statistico degli anni '1970 "Analisi dei dati esplorativiÈ ormai affermato come un processo importante nell'analisi dei dati, con circa 2 citazioni (Tukey, John W. Analisi dei dati esplorativiVol. 2. 1977.).
Preparazione dei dati
BBBP di Molecule Net viene utilizzato per i dati del campione. Per quanto riguarda la penetrazione della barriera ematoencefalica, i dati sono una sintesi di "penetrazione" come XNUMX e "non penetrazione" come XNUMX.
riferimenti:https://pubs.acs.org/doi/10.1021/ci300124c
Preparazione dei dati
import numpy as np
import pandas as pd
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, PandasTools, Descriptors
pd.set_option('display.max_columns',250)
print('rdkit version: ',rdBase.rdkitVersion) #rdkit version: 2019.03.4
bbbp = pd.read_csv('./BBBP.csv',index_col=0)
# smilesからmolオブジェクトを作成し、DataFrameに格納
PandasTools.AddMoleculeColumnToFrame(bbbp,'smiles')
# molオブジェクトを作れなかった行の削除
bbbp = bbbp.dropna()
bbbp.info()Int64 Indice: 2039 voci, da 1 a 2053 Colonne dati (totale 4 colonne): nome 2039 oggetto non nullo p_np 2039 non nullo int64 sorrisi 2039 oggetto non nullo ROMol 2039 oggetto non nullo dtypes: int64 (1), oggetto (3 )) utilizzo della memoria: 79.6+ KB
Una vista a volo d'uccello del set di dati
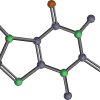
Innanzitutto, controlla l'elenco di quale tipo di struttura chimica è inclusa.
# 化学構造を一覧で表示
PandasTools.FrameToGridImage(bbbp[:18], column='ROMol', legendsCol='name', molsPerRow=6, subImgSize=(150,150))
# ターゲットp-npの分布を確認
bbbp.p_np.value_counts()1 1560 0 Nome: p_np, dtype: int479
Cerca la correlazione con la variabile oggettiva dal descrittore del composto
Poiché non è possibile ottenere una panoramica semplicemente guardandola, creare e aggregare descrittori e ottenere una vista a volo d'uccello del set di dati.
I descrittori dei composti sono riassunti di seguito.
# 記述子を生成する
for i,j in Descriptors.descList:
bbbp[i] = bbbp.ROMol.map(j)
# 要約統計量の表示
bbbp.describe()
# データセット全体における構造記述子の分布
import matplotlib.pyplot as plt
import math
list = ["p_np", "RingCount", "NumAromaticRings", "NumAromaticCarbocycles", "NumAromaticHeterocycles", "NumSaturatedCarbocycles", "NumSaturatedHeterocycles", "NumSaturatedRings"]
fig = plt.figure(figsize=(15,7))
for i, c in enumerate(list):
ax = fig.add_subplot(
math.ceil(len(list) / 4), 4, i + 1)
# plot the continent on these axes
sns.countplot(x=c, data=bbbp, ax=ax)
ax.set_title(c)
fig.tight_layout()
plt.show()
# targetによる構造記述子の分布の違いを可視化
list = ["RingCount", "NumAromaticRings", "NumAromaticCarbocycles", "NumAromaticHeterocycles", "NumSaturatedCarbocycles", "NumSaturatedHeterocycles", "NumSaturatedRings"]
fig = plt.figure(figsize=(15,7))
for i, c in enumerate(list):
ax = fig.add_subplot(
math.ceil(len(list) / 4), 4, i + 1)
# plot the continent on these axes
sns.countplot(x=c, data=bbbp, ax=ax, hue="p_np")
ax.set_title(c)
fig.tight_layout()
plt.show()
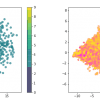
# logPと分子量でplotしてみる
import seaborn as sns
sns.scatterplot(x="MolWt", y="MolLogP", data=bbbp,
hue="p_np",
alpha=0.5)
Poiché è la permeabilità della barriera emato-encefalica, fattori importanti erano quasi noti, ma si può vedere che l'idrofobicità (logP) e il peso molecolare (MolWt) sono importanti per la permeabilità della membrana.
I seguenti sono stati utili come metodi per l'analisi esplorativa generale dei dati ( Come scegliere e disegnare il metodo di visualizzazione corretto nell'analisi esplorativa dei dati ).Sembra opportuno applicarlo all'aggregazione dei descrittori.









discussione
Elenco dei commenti
Non ci sono ancora commenti