Calcule el descriptor molecular y la huella digital de SMILES y guárdelo en el marco de datos [Python, RDKit]
Cómo crear un marco de datos que contenga descriptores moleculares y huellas dactilares de SMILES de un conjunto de datos compuesto con RDKit. Incluso si intenté hacer mi propio modelo de aprendizaje automático / QSAR, tropecé con la creación de descriptores moleculares y huellas digitales, por lo que los resumiré a continuación.
Importancia de almacenar en el marco de datos
Puede crear un tipo de lista solo para ponerlo en aprendizaje automático, pero el uso de un marco de datos facilita lo siguiente.
- Una vista panorámica del conjunto de datos compuesto basado en el descriptor / huella digital creado
- Procesamiento previo de datos, como manejo de valores perdidos y reducción de dimensiones
Además, en RDKit, SMILES se convierte una vez en un objeto mol para calcular el descriptor, pero incluso si hay algo que no se pudo convertir bien en ese momento, el marco de datos es más fácil de manejar.
Intenta practicar
Preparación
Para los datos de muestra, utilizaremos SMIELS de BBBP (conjunto de datos de penetración de la barrera hematoencefálica) de MoleculeNet.
El objeto mol de RDKit se almacena en una columna llamada ROMol, así que cree un descriptor basado en esto.
Referencia: Lista de conjuntos de datos compuestos
import numpy as np
import pandas as pd
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, PandasTools, Descriptors
from rdkit.Chem.Draw import IPythonConsole
print('rdkit version: ',rdBase.rdkitVersion) # rdkit version: 2019.03.4
# 下準備
# データセットの読み込み
df = pd.read_csv("BBBP.csv")
# dfのSMILES列を参照してMolオブジェクト列をデータフレームに加える
PandasTools.AddMoleculeColumnToFrame(df,'smiles')
# Molオブジェクトが作成できたか確認
print(df.shape)
print(df.isnull().sum()) (2050, 4) num 0 nombre 0 p_np 0 sonrisas 0 ROMol 11 dtype: int64
Se muestra el error "La valencia explícita para el átomo n. ° 1 N, 4, es mayor que la permitida" porque había una molécula (ión, etc.) con una valencia anormal ("4 átomos en N"). El valor está por encima del permitido valor. ")Ninguno fue devuelto al ROMol de tal molécula, y había 11 de esas SONRISAS aquí.
Puede tratar con ellos uno por uno, pero si el número es pequeño, es rápido eliminarlos por el momento.Por lo tanto, use isnull (). Sum () para verificar si faltan valores en la columna ROMol, y elimine esas filas si las hay.
Referencia: Solución de problemas de lectura de datos compuestos
# ROMolが作成できなかったものを確認
print(df[df.ROMol.isnull()])
# 欠損行の除去
df = df.dropna() 
Si ve "ADVERTENCIA: no eliminar átomos de hidrógeno sin vecinos", probablemente se deba a que los datos contienen sales. RDKit guarda H de forma predeterminada, por lo que si hay una H (como sal) que no está vinculada al vecino, dicha H no se puede eliminar y le advierte.
Creando un descriptor molecular
La función Mapa es útil para aplicar una función a un objeto en cada fila de un marco de datos.
Dado que los nombres y funciones de los descriptores se enumeran en "Descriptors.descList" de RDKit, lleva un poco de tiempo, pero pude calcular todo a la vez con la función for y la función map y devolverlo al marco de datos.
for i,j in Descriptors.descList:
df[i] = df.ROMol.map(j)
df.shape
# (2039, 205)
df.head()
Se han agregado descriptores para 201 columnas.
Al aplicar la variable obtenida a scikit-learn o un marco de aprendizaje profundo, puede obtener el error "Error de valor: la entrada contiene NaN, infinito o un valor demasiado grande para dtype ('float64')". Estaba bien si hice lo siguiente .
for i,j in Descriptors.descList:
df[i] = df['ROMol'].map(j)
df['Ipc'] = [Descriptors.Ipc(mol, avg=True) for mol in df['ROMol']] Parece que la causa es que una parte del valor del descriptor "IPC" se crea tan grande como infinito.
Referencia: # 12 Qué hacer si el valor de IPC del descriptor RDKit 2D es muy grande
Referencia: Lista de descriptores moleculares
Creando una huella dactilar
Se puede calcular rápidamente usando la función de aplicación, pero parece que la lista de huellas digitales se almacena en una columna.
Dado que la huella digital se guarda en el formato de objeto BitVect explícito, tomó algún tiempo almacenar cada valor en una columna.
# 下準備
df = pd.read_csv("BBBP.csv")
PandasTools.AddMoleculeColumnToFrame(df,'smiles')
df = df.dropna()
# 1列にfingerprintのリストを追加する場合
df['FP'] = df.apply(lambda x: AllChem.GetMorganFingerprintAsBitVect(x.ROMol, 2, 1024), axis=1)
# fingerprintの各値を各列に格納する場合
# 個別に01をデータフレームに格納する
FP = [AllChem.GetMorganFingerprintAsBitVect(mol, 2, 1024) for mol in df.ROMol]
df_FP = pd.DataFrame(np.array(FP))
# フィンガープリントをもとのデータフレームに結合
df_FP.index = df.index
df = pd.concat([df, df_FP], axis=1)

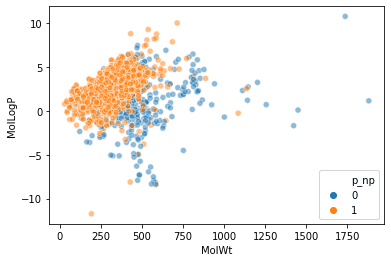









discusión
Lista de comentarios
Aún no hay comentarios