Chainer로 간단한 QSAR 모델을 시험해 본다 [화합물의 혈액뇌 장벽 투과성을 예측한다]
QSAR(정량적 구조 활성 상관: Quantitative Structure-Activity Relationship)이란, 화학물질의 구조와 그 생리 활성(독성·효소에의 결합능·의약품으로서의 작용성 등)의 통계적인 상관관계를 좋아.방대한 화학 물질의 실험 데이터 세트를 기반으로 상관 관계에서 화합물의 성능을 예측할 수 있습니다.
이번은 일본제의 딥 러닝 프레임워크인 Chainer를 이용해 「화합물의 혈액뇌 장벽 투과성을 예측한다」간단한 QSAR 모델이 작성해, 테스트 세트에 대한 성능을 검증해 보겠습니다.
예측 대상 및 데이터
데이터에는 MoleculeNet의 BBBP를 이용.데이터의 부감은 이전에 실시한 이하를 참조.
화합물의 혈액뇌 장벽 투과성에 대해 「투과성 있음(penetration)」을 XNUMX, 「투과성 없음(non-penetration)」을 XNUMX으로 정리한 데이터입니다.
모델 만들기
환경
from rdkit import rdBase
import chainer
print('rdkit version: ',rdBase.rdkitVersion)
chainer.print_runtime_info()rdkit version: 2019.03.4 Platform: Linux-5.0.0-37-generic-x86_64-with-debian-buster-sid Chainer: 6.2.0 NumPy: 1.17.4 CuPy: CuPy Version : 6.2.0 CUDA Root : /local/cuda CUDA Build Version : 10010 CUDA Driver Version : 10010 CUDA Runtime Version : 10010 cuDNN Build Version : 7500 cuDNN Version : 7605 NCCL Build Version : 2402 NCCL Run
chainer.print_runtime_info()에서 사용하고 있는 Chainer, Numpy, Cupy의 버전을 확인할 수 있습니다.
모델 구축
import numpy as np
import pandas as pd
from rdkit import Chem
from rdkit.Chem import Draw, PandasTools, Descriptors
# データの読み込み
df = pd.read_csv('BBBP.csv',index_col=0)
# smilesからmolファイルを生成し、データフレーム中に加える
PandasTools.AddMoleculeColumnToFrame(df, smilesCol = 'smiles')
# molができなかった行を削除する
df = df.dropna()
# molファイルから化合物記述子を算出する
for i,j in Descriptors.descList:
df[i] = df['ROMol'].map(j)
df['Ipc'] = [Descriptors.Ipc(mol, avg=True) for mol in df['ROMol']]
# chainer用にデータ型を変換
x = df.iloc[:,4:].values.astype('float32')
y = df['p_np'].values.astype('int32')
indices = np.array(range(x.shape[0])) # train_test_split後も列番号を保持しておく
# train, test, valに分割
from sklearn.model_selection import train_test_split
x_train, x_test, y_train, y_test, indices_train, indices_test = train_test_split(x, y, indices, test_size=0.05, random_state=123)
# 説明変数の標準化
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
scaler.fit(x_train)
x_train= scaler.transform(x_train)
x_test = scaler.transform(x_test)
print(type(x_train), x_train.shape, type(y_train), y_train.shape)
print(type(x_test), x_test.shape, type(y_test), y_test.shape)(1937, 200) (1937,) (102, 200) (102,)
mol 객체로부터의 기술자 작성에 대해서는 이쪽을 참조.
SMILES에서 분자 기술자와 지문을 계산하여 데이터 프레임에 저장하는 [Python, RDKit]
Chainer의 기본 사용법은 공식 튜토리얼이 매우 충실합니다.
딥 러닝 입문 Chainer 튜토리얼
# 説明変数と目的変数のセットで使えるように変換する
from chainer.datasets import TupleDataset
train = TupleDataset(x_train, y_train)
test = TupleDataset(x_test, y_test)
# イテレータの準備
from chainer.iterators import SerialIterator
train_iter = SerialIterator(train, batch_size=64, repeat=True, shuffle=True)
test_iter = SerialIterator(test, batch_size=64, shuffle=False, repeat=False)
# ニューラルネットワークの作成
# 3層のmulti layer perceptron(MLP)
import chainer.links as L
import chainer.functions as F
from chainer import Chain
from chainer import optimizers, training
from chainer.training import extensions
class MLP(chainer.Chain):
def __init__(self):
super().__init__()
with self.init_scope():
self.fc1 = L.Linear(None, 100)
self.fc2 = L.Linear(None, 20)
self.fc3 = L.Linear(None, 2)
def forward(self, x):
h = F.relu(self.fc1(x))
h = F.relu(self.fc2(h))
h = self.fc3(h)
return h
# ネットワークをClassifierでラップしする
# (目的関数(デフォルトはsoftmax交差エントロピー)の計算し、損失を返す)
predictor = MLP()
net = L.Classifier(predictor)
# 最適化手法を選択して、オプティマイザを作成する
optimizer = optimizers.MomentumSGD(lr=0.1).setup(net)
# アップデータにイテレータとオプティマイザを渡す
updater = training.StandardUpdater(train_iter, optimizer, device=-1)
trainer = training.Trainer(updater, (50, 'epoch'), out='/results/')from chainer.training import extensions
trainer.extend(extensions.LogReport(trigger=(5, 'epoch'), log_name='log'))
trainer.extend(extensions.snapshot(filename='snapshot_epoch-{.updater.epoch}'))
trainer.extend(extensions.dump_graph('main/loss'))
trainer.extend(extensions.Evaluator(test_iter, net, device=-1), name='val')
trainer.extend(extensions.PrintReport(['epoch', 'iteration', 'main/loss', 'main/accuracy', 'val/main/loss', 'val/main/accuracy', 'fc1/W/data/mean', 'elapsed_time']))
trainer.extend(extensions.PlotReport(['fc1/W/grad/mean'], x_key='epoch', file_name='mean.png'))
trainer.extend(extensions.PlotReport(['main/loss', 'val/main/loss'], x_key='epoch', file_name='loss.png'))
trainer.extend(extensions.PlotReport(['main/accuracy', 'val/main/accuracy'], x_key='epoch', file_name='accuracy.png'))
trainer.extend(extensions.ParameterStatistics(net.predictor.fc1, {'mean': np.mean}, report_grads=True))
trainer.run()from IPython.display import Image, display
display(Image(filename='results/accuracy.png'))
나름의 정밀도는 나올 것 같지만, 학습이 진행되어도 테스트 세트에 대한 accuracy가 그다지 개선하고 있지 않다…
추론
# 学習したモデルで推論してみる
with chainer.using_config('train', False), chainer.using_config('enable_backprop', False):
y_pred = predictor(x_test)
# 推論結果の確認
print('accuracy', F.accuracy(y_pred, y_test)) # accuracy variable(0.88235295)
from sklearn.metrics import confusion_matrix
confusion_matrix(y_test, y_pred.data.argmax(axis=1))array([[21, 7], [ 5, 69]])
accuracy는 그림에서 알 수 있듯이 값.혼동 행렬에서 클래스 분류의 정밀도를 평가해 보면 위양성, 위음성도 있지만, 한편으로 편향된 분류로 정밀도를 벌고 있는 것도 아닐 것 같다.
# 一部予測結果を見てみる
for i in range(int(len(y_pred)/10)):
print('No.', indices_test[i])
print('label:', y_test[i])
print('pred :', np.argmax(y_pred[i].array))
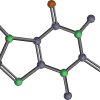
img = Draw.MolToImage(df.ROMol[indices_test[i]])
display(img)
이미지는 출력의 일부입니다만, 눈으로도 판별할 수 있을 것 같은 것은 제대로 정답이 되어 있습니다.실수한 것을 조사하는 것도 즐거울 것 같습니다.










토론
코멘트 목록
아직 코멘트가 없습니다.