Bereken de moleculaire descriptor en vingerafdruk van SMILES en sla deze op in het dataframe [Python, RDKit]
Hoe maak je een dataframe met moleculaire descriptoren en vingerafdrukken van SMILES van een samengestelde dataset met RDKit. Zelfs als ik probeerde mijn eigen QSAR / machine learning-model te maken, stuitte ik op het maken van moleculaire descriptoren en vingerafdrukken, dus ik zal ze hieronder samenvatten.
Betekenis van het opslaan in dataframe
U kunt een lijsttype maken om het in machine learning te plaatsen, maar door een dataframe te gebruiken, is het gemakkelijker om het volgende te doen.
- Een vogelvlucht van de samengestelde dataset op basis van de gemaakte descriptor / vingerafdruk
- Voorverwerking van gegevens, zoals afhandeling van ontbrekende waarden en verkleining van afmetingen
Ook wordt in RDKit SMILES eenmaal geconverteerd naar een mol-object om de descriptor te berekenen, maar zelfs als er iets is dat op dat moment niet goed kon worden geconverteerd, is het dataframe gemakkelijker te hanteren.
Probeer te oefenen
Voorbereiding
Voor voorbeeldgegevens zullen we SMIELS van MoleculeNet's BBBP (bloed-hersenbarrière penetratie dataset) gebruiken.
Het RDKit mol-object wordt opgeslagen in een kolom genaamd ROMol, dus maak op basis hiervan een descriptor.
Referentie: lijst met samengestelde datasets
import numpy as np
import pandas as pd
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, PandasTools, Descriptors
from rdkit.Chem.Draw import IPythonConsole
print('rdkit version: ',rdBase.rdkitVersion) # rdkit version: 2019.03.4
# 下準備
# データセットの読み込み
df = pd.read_csv("BBBP.csv")
# dfのSMILES列を参照してMolオブジェクト列をデータフレームに加える
PandasTools.AddMoleculeColumnToFrame(df,'smiles')
# Molオブジェクトが作成できたか確認
print(df.shape)
print(df.isnull().sum()) (2050, 4) num 0 naam 0 p_np 0 glimlachen 0 ROMol 11 dtype: int64
De fout 'Expliciete valentie voor atoom # 1 N, 4, is groter dan toegestaan' wordt weergegeven omdat er een molecuul (ion, enz.) Was met een abnormale valentie ('4 atomen in N'). De waarde ligt boven de toegestane waarde waarde. ")Geen enkele was teruggekeerd naar de ROMol van zo'n molecuul, en er waren hier 11 van zulke GLIMLACHEN.
Je kunt ze een voor een afhandelen, maar als het aantal klein is, kun je ze voorlopig snel verwijderen.Gebruik daarom isnull (). Sum () om te controleren op ontbrekende waarden in de ROMol-kolom en verwijder die rijen, indien aanwezig.
Referentie: problemen oplossen met het lezen van samengestelde gegevens
# ROMolが作成できなかったものを確認
print(df[df.ROMol.isnull()])
# 欠損行の除去
df = df.dropna() 
Als je "WAARSCHUWING: geen waterstofatomen verwijderen zonder buren" ziet, komt dat waarschijnlijk doordat de data zouten bevat. RDKit slaat H standaard op, dus als er een H is (zoals salt) die niet aan de buur is gebonden, kan zo'n H niet worden verwijderd en wordt je gewaarschuwd.
Een moleculaire descriptor maken
De kaartfunctie is handig om een functie toe te passen op een object in elke rij in een dataframe.
Aangezien de descriptornamen en functies worden vermeld in "Descriptors.descList" van RDKit, kost het wat tijd, maar ik kon alles tegelijk berekenen met de functie for en map en deze terugzetten naar het dataframe.
for i,j in Descriptors.descList:
df[i] = df.ROMol.map(j)
df.shape
# (2039, 205)
df.head()
Er zijn descriptoren voor 201 kolommen toegevoegd.
Wanneer u de verkregen variabele toepast op scikit-learn of een deep learning-framework, krijgt u mogelijk de fout "Waardefout: invoer bevat NaN, oneindig of een waarde die te groot is voor dtype ('float64')". Was prima als ik het volgende deed .
for i,j in Descriptors.descList:
df[i] = df['ROMol'].map(j)
df['Ipc'] = [Descriptors.Ipc(mol, avg=True) for mol in df['ROMol']] Het lijkt erop dat de oorzaak is dat een deel van de descriptor "IPC" -waarde zo groot als oneindig wordt gecreëerd.
Referentie: # 12 Wat te doen als de IPC-waarde van de RDKit 2D-descriptor erg groot is
Referentie: lijst met moleculaire descriptoren
Een vingerafdruk maken
Het kan snel worden berekend met de functie Apply, maar het lijkt erop dat de lijst met vingerafdrukken in één kolom is opgeslagen.
Omdat de vingerafdruk wordt opgeslagen in de indeling van een expliciet BitVect-object, duurde het even voordat elke waarde in één kolom was opgeslagen.
# 下準備
df = pd.read_csv("BBBP.csv")
PandasTools.AddMoleculeColumnToFrame(df,'smiles')
df = df.dropna()
# 1列にfingerprintのリストを追加する場合
df['FP'] = df.apply(lambda x: AllChem.GetMorganFingerprintAsBitVect(x.ROMol, 2, 1024), axis=1)
# fingerprintの各値を各列に格納する場合
# 個別に01をデータフレームに格納する
FP = [AllChem.GetMorganFingerprintAsBitVect(mol, 2, 1024) for mol in df.ROMol]
df_FP = pd.DataFrame(np.array(FP))
# フィンガープリントをもとのデータフレームに結合
df_FP.index = df.index
df = pd.concat([df, df_FP], axis=1)

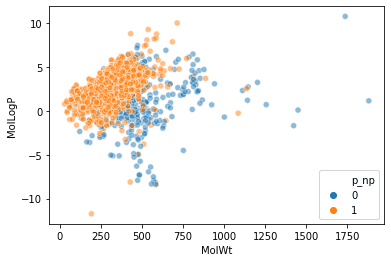









discussie
Lijst met opmerkingen
Er zijn nog geen reacties