คำนวณตัวบอกลักษณะโมเลกุลและลายนิ้วมือจาก SMILES และจัดเก็บไว้ใน data frame [Python, RDKit]
นี่คือวิธีการสร้างกรอบข้อมูลที่ประกอบด้วยตัวบอกลักษณะโมเลกุลและลายนิ้วมือจากชุดข้อมูลผสม SMILES ด้วย RDKit ฉันพยายามสร้าง QSAR/โมเดลแมชชีนเลิร์นนิงของฉันเอง แต่ฉันสะดุดนิดหน่อยกับการสร้างตัวบอกลักษณะระดับโมเลกุลและลายนิ้วมือ ดังนั้นฉันจะสรุปไว้ด้านล่าง
ความสำคัญของการจัดเก็บในกรอบข้อมูล
หากเป็นเพียงการเรียนรู้ของเครื่อง คุณสามารถสร้างเป็นประเภทรายการได้ แต่การทำให้เป็นเฟรมข้อมูลจะทำให้ทำสิ่งต่อไปนี้ได้ง่ายขึ้น
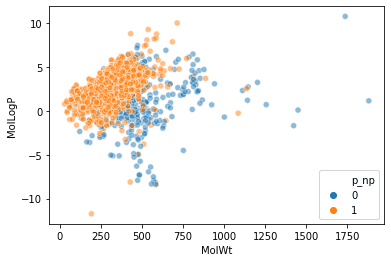
- มุมมองจากมุมสูงของชุดข้อมูลผสมตามคำอธิบาย/ลายนิ้วมือที่สร้างขึ้น
- การประมวลผลข้อมูลล่วงหน้า เช่น การจัดการค่าที่ขาดหายไปและการลดขนาด
นอกจากนี้ RDKit จะแปลง SMILES เป็นวัตถุ mol ในขั้นแรกเพื่อคำนวณตัวอธิบาย แต่เฟรมข้อมูลจะจัดการได้ง่ายกว่าแม้ว่าจะมีสิ่งที่ไม่สามารถแปลงได้ดีก็ตาม
ลองดูสิ
การตระเตรียม
สำหรับข้อมูลตัวอย่าง เราจะใช้ SMIELS ของ MoleculeNet (ชุดข้อมูลการเจาะสิ่งกีดขวางระหว่างเลือดและสมอง)
วัตถุ RDKit mol ถูกจัดเก็บไว้ในคอลัมน์ชื่อ ROMol ดังนั้นให้สร้างคำอธิบายตามสิ่งนี้
import numpy as np
import pandas as pd
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, PandasTools, Descriptors
from rdkit.Chem.Draw import IPythonConsole
print('rdkit version: ',rdBase.rdkitVersion) # rdkit version: 2019.03.4
# 下準備
# データセットの読み込み
df = pd.read_csv("BBBP.csv")
# dfのSMILES列を参照してMolオブジェクト列をデータフレームに加える
PandasTools.AddMoleculeColumnToFrame(df,'smiles')
# Molオブジェクトが作成できたか確認
print(df.shape)
print(df.isnull().sum()) (2050, 4) จำนวน 0 ชื่อ 0 p_np 0 ยิ้ม 0 ROMol 11 dtype: int64
ข้อผิดพลาด "ความจุชัดเจนสำหรับอะตอม # 1 N, 4 มากกว่าที่อนุญาต" ปรากฏขึ้น แต่สิ่งนี้เกิดจากโมเลกุล (เช่น ไอออน) ที่มีเวเลนซ์ผิดปกติ ("อะตอม # 4 N, 11 มากกว่าที่อนุญาต ") ค่าเกินค่าที่ยอมรับได้”)ไม่มีการส่งกลับใน ROMol สำหรับโมเลกุลดังกล่าว และมี XNUMX SMILES ดังกล่าวที่นี่
คุณสามารถจัดการกับพวกมันทีละตัว แต่ถ้าจำนวนน้อย การลบพวกมันออกชั่วคราวจะเร็วกว่าดังนั้น ใช้ isnull().sum() เพื่อตรวจสอบว่ามีค่าขาดหายไปในคอลัมน์ ROMol หรือไม่ และหากมี ให้ลบแถวออก
อ้างอิง: การแก้ไขปัญหาการโหลดข้อมูลผสม
# ROMolが作成できなかったものを確認
print(df[df.ROMol.isnull()])
# 欠損行の除去
df = df.dropna() 
หากคุณเห็น "คำเตือน: ห้ามกำจัดอะตอมของไฮโดรเจนโดยไม่มีเพื่อนบ้าน" อาจเป็นเพราะข้อมูลมีเกลืออยู่ ตามค่าเริ่มต้น RDKit จะบันทึกข้อมูลโดยนำ H ออก ดังนั้นหากมี H (เช่น เกลือ) ที่ไม่ผูกมัดกับเพื่อนบ้าน ก็จะไม่สามารถลบ H ดังกล่าวออกได้และจะมีการเตือน
การสร้างตัวบ่งชี้ระดับโมเลกุล
ฟังก์ชัน Map มีประโยชน์สำหรับการใช้ฟังก์ชันกับแต่ละแถวในดาต้าเฟรม
เนื่องจากชื่อ descriptor และฟังก์ชันแสดงอยู่ใน "Descriptors.descList" ของ RDKit จึงใช้เวลาพอสมควร แต่ฉันสามารถทำการคำนวณแบบกลุ่มด้วยฟังก์ชัน for และฟังก์ชัน map และส่งกลับไปยัง data frame
for i,j in Descriptors.descList:
df[i] = df.ROMol.map(j)
df.shape
# (2039, 205)
df.head()
เพิ่มคำอธิบายสำหรับ 201 คอลัมน์
เมื่อใช้ตัวแปรที่ได้รับกับ scikit-learn หรือกรอบการเรียนรู้เชิงลึก คุณอาจได้รับข้อผิดพลาด "ValueError: Input ประกอบด้วย NaN, infinity หรือค่าที่ใหญ่เกินไปสำหรับ dtype ('float64')" แต่ในกรณีนั้น ไม่เป็นไรถ้า ฉันทำ:
for i,j in Descriptors.descList:
df[i] = df['ROMol'].map(j)
df['Ipc'] = [Descriptors.Ipc(mol, avg=True) for mol in df['ROMol']] ดูเหมือนว่าสาเหตุคือค่าขนาดใหญ่ที่ไม่สิ้นสุดถูกสร้างขึ้นในค่า "IPC" ของส่วนหนึ่งของตัวอธิบาย
อ้างอิง: #12 จะทำอย่างไรเมื่อค่า IPC ในตัวบอก RDKit 2D มีขนาดใหญ่มาก
อ้างอิง: รายการตัวบ่งชี้ระดับโมเลกุล
สร้างลายนิ้วมือ
สามารถคำนวณได้อย่างรวดเร็วโดยใช้ฟังก์ชันนำไปใช้ แต่ดูเหมือนว่ารายการลายนิ้วมือจะถูกจัดเก็บไว้ในคอลัมน์เดียว
ลายนิ้วมือถูกจัดเก็บไว้ในรูปแบบวัตถุ ExplicitBitVect ดังนั้นการจัดเก็บแต่ละค่าในแถวจึงค่อนข้างยุ่งยาก
# 下準備
df = pd.read_csv("BBBP.csv")
PandasTools.AddMoleculeColumnToFrame(df,'smiles')
df = df.dropna()
# 1列にfingerprintのリストを追加する場合
df['FP'] = df.apply(lambda x: AllChem.GetMorganFingerprintAsBitVect(x.ROMol, 2, 1024), axis=1)
# fingerprintの各値を各列に格納する場合
# 個別に01をデータフレームに格納する
FP = [AllChem.GetMorganFingerprintAsBitVect(mol, 2, 1024) for mol in df.ROMol]
df_FP = pd.DataFrame(np.array(FP))
# フィンガープリントをもとのデータフレームに結合
df_FP.index = df.index
df = pd.concat([df, df_FP], axis=1)









ดิสโก้
รายการความคิดเห็น
ยังไม่มีความเห็น