Trực quan hóa mạng lưới thần kinh trong R
Gói trực quan mạng thần kinh trong R
Hàm mạng thần kinh có thể trực quan hóa đồ thị tính toán với hàm plot() làm tiêu chuẩn. Lưu ý bên dưới cách trực quan hóa biểu đồ tính toán khi sử dụng các gói mạng thần kinh khác thiếu các tính năng như chức năng mạng thần kinh.
- hàm plot.nn
- chức năng cốt truyện
sự chuẩn bị
Dữ liệu mẫu sử dụng mống mắt
Tạo người học
d=iris d$Species <- as.factor(d$Species) #train_test_split set.seed(0) sample <- sample.int(n = nrow(d), size = floor(0.80*nrow(d)), replace = F) train <- d[sample, ] test <- d[-sample, ] summary(train) #nnet library(nnet) nn1=nnet(Species~., size=5, data=train) pred_nn1 <- bảng dự đoán(nn1, test,type="class")(test$Species,pred_nn1)

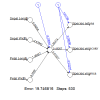
hình dung nnet
Trong mỗi trường hợp, màu biểu thị dương hoặc âm và độ dày biểu thị độ lớn của giá trị số.
hàm plot.nn
nguồn ("http://hosho.ees.hokudai.ac.jp/~kubo/log/2007/img07/plot.nn.txt") cốt truyện.nn(nn1)

chức năng cốt truyện.nnet
thư viện install.packages("NeuralNetTools")(NeuralNetTools) plotnet(nn1)
Nhân tiện, trong hàm neuralnet
thư viện(dấu mũ) tmp <- dummyVars(~.,data=train) dummy <- as.data.frame(predict(tmp, train)) thư viện("neuralnet") f = Species.setosa + Species.versicolor + Species. virginica~ Sepal.Length + Sepal.Width + Petal.Length + Petal.Width nn2 <- neuralnet(công thức = f, dữ liệu = giả) cốt truyện (nn2)
Khi có nhiều biến, việc xem hình ảnh trực quan sẽ dễ dàng hơn khi sử dụng chức năng biểu đồ ngang. Chức năng plotnet rất dễ sử dụng vì nó có thể trực quan hóa không chỉ nnet mà còn cả các mạng thần kinh được tạo bằng RSNNS và dấu mũ, đồng thời có nhiều ứng dụng.









thảo luận
Danh sách các bình luận
Chưa có bất kỳ bình luận nào