从SMILES计算分子描述符和指纹并将它们存储在数据框中[Python,RDKit]
如何使用RDKit从化合物数据集中的SMILES创建包含分子描述符和指纹的数据框。 即使我尝试建立自己的QSAR /机器学习模型,也偶然发现了创建分子描述符和指纹的方法,因此下面将对其进行总结。
在数据框中存储的意义
您可以创建列表类型只是为了将其放入机器学习中,但是将其设为数据框可以使执行以下操作更加容易。
- 基于创建的描述符/指纹的化合物数据集的鸟瞰图
- 数据预处理,例如缺失值处理和降维
同样,在RDKit中,SMILES曾经被转换为mol对象以计算描述符,但是即使那时有些东西不能很好地转换,数据帧也更易于处理。
尝试练习
准备
对于样本数据,我们将使用来自 MoleculeNet 的 BBBP(血脑屏障穿透数据集)的 SMIELS。
RDKit mol对象存储在名为ROMol的列中,因此请基于此创建描述符。
import numpy as np
import pandas as pd
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, PandasTools, Descriptors
from rdkit.Chem.Draw import IPythonConsole
print('rdkit version: ',rdBase.rdkitVersion) # rdkit version: 2019.03.4
# 下準備
# データセットの読み込み
df = pd.read_csv("BBBP.csv")
# dfのSMILES列を参照してMolオブジェクト列をデータフレームに加える
PandasTools.AddMoleculeColumnToFrame(df,'smiles')
# Molオブジェクトが作成できたか確認
print(df.shape)
print(df.isnull().sum()) (2050,4)num 0名称0 p_np 0微笑0 ROMol 11 dtype:int64
由于存在一个分子(离子等)具有异常的化合价(“ N中有1个原子”),所以显示错误“原子#4 N,4的显着化合价大于允许值”。该值高于允许值价值。 ”)没有一个返回到这种分子的ROMol,这里有11个这样的SMILES。
您可以一对一地处理它们,但是如果数量很少,可以暂时将其删除。因此,请使用notull()。Sum()检查ROMol列中的缺失值,并删除这些行(如果有的话)。
# ROMolが作成できなかったものを確認
print(df[df.ROMol.isnull()])
# 欠損行の除去
df = df.dropna() 
如果看到“警告:没有邻居就不要去除氢原子”,这可能是因为数据包含盐。 RDKit默认情况下会保存H,因此,如果存在不与邻居绑定的H(例如盐),则无法删除该H并发出警告。
创建分子描述符
映射功能对于将功能应用于数据框中每一行的对象很有用。
由于描述符名称和函数在RDKit的“ Descriptors.descList”中列出,因此需要花费一些时间,但是我能够使用for函数和map函数一次计算所有值并将其返回到数据帧。
for i,j in Descriptors.descList:
df[i] = df.ROMol.map(j)
df.shape
# (2039, 205)
df.head()
添加了201列的描述符。
将获得的变量应用于scikit-learn或深度学习框架时,您可能会收到错误“值错误:输入包含NaN,无穷大或对于dtype('float64')而言太大的值。” 。
for i,j in Descriptors.descList:
df[i] = df['ROMol'].map(j)
df['Ipc'] = [Descriptors.Ipc(mol, avg=True) for mol in df['ROMol']] 似乎原因是描述符“ IPC”值的一部分被创建为无穷大。
参考:#12如果RDKit 2D描述符的IPC值很大,该怎么办
参考:分子描述符列表
创建指纹
可以使用apply函数快速计算出来,但似乎指纹列表存储在一列中。
由于指纹以Explicit BitVect对象的格式保存,因此花了一些时间将每个值存储在一列中。
# 下準備
df = pd.read_csv("BBBP.csv")
PandasTools.AddMoleculeColumnToFrame(df,'smiles')
df = df.dropna()
# 1列にfingerprintのリストを追加する場合
df['FP'] = df.apply(lambda x: AllChem.GetMorganFingerprintAsBitVect(x.ROMol, 2, 1024), axis=1)
# fingerprintの各値を各列に格納する場合
# 個別に01をデータフレームに格納する
FP = [AllChem.GetMorganFingerprintAsBitVect(mol, 2, 1024) for mol in df.ROMol]
df_FP = pd.DataFrame(np.array(FP))
# フィンガープリントをもとのデータフレームに結合
df_FP.index = df.index
df = pd.concat([df, df_FP], axis=1)

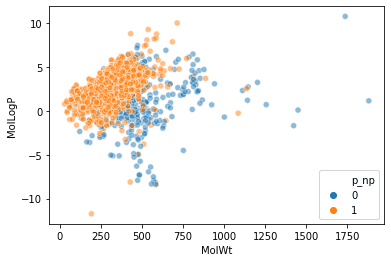









讨论
评论清单
暂无任何评论