احسب الواصف الجزيئي وبصمة الإصبع من SMILES وقم بتخزينها في إطار البيانات [Python ، RDKit]
كيفية إنشاء إطار بيانات يحتوي على واصفات جزيئية وبصمات أصابع من SMILES لمجموعة بيانات مركبة باستخدام RDKit. حتى لو حاولت إنشاء نموذج QSAR / التعلم الآلي الخاص بي ، فقد عثرت على واصفات جزيئية وبصمات أصابع ، لذلك سألخصها أدناه.
أهمية التخزين في إطار البيانات
يمكنك إنشاء نوع قائمة فقط لوضعه في التعلم الآلي ، ولكن استخدام إطار البيانات يجعل من السهل القيام بما يلي.
- عرض شامل لمجموعة البيانات المركبة بناءً على الواصف / بصمة الإصبع التي تم إنشاؤها
- المعالجة المسبقة للبيانات مثل معالجة القيمة المفقودة وتقليل الأبعاد
بالإضافة إلى ذلك ، يقوم RDKit مرة واحدة بتحويل SMILES إلى كائن mol من أجل حساب الواصف ، ولكن حتى إذا كان هناك شيء لا يمكن تحويله جيدًا في ذلك الوقت ، فإن إطار البيانات يكون أسهل في التعامل معه.
حاول أن تتدرب
تحضير
للحصول على بيانات العينة ، سوف نستخدم SMIELS من MoleculeNet's BBBP (مجموعة بيانات اختراق الحاجز الدموي الدماغي).
يتم تخزين كائن RDKit mol في عمود يسمى ROMol ، لذلك قم بإنشاء واصف بناءً على هذا.
المرجع: قائمة مجموعات البيانات المركبة
import numpy as np
import pandas as pd
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, PandasTools, Descriptors
from rdkit.Chem.Draw import IPythonConsole
print('rdkit version: ',rdBase.rdkitVersion) # rdkit version: 2019.03.4
# 下準備
# データセットの読み込み
df = pd.read_csv("BBBP.csv")
# dfのSMILES列を参照してMolオブジェクト列をデータフレームに加える
PandasTools.AddMoleculeColumnToFrame(df,'smiles')
# Molオブジェクトが作成できたか確認
print(df.shape)
print(df.isnull().sum()) (2050، 4) num 0 name 0 p_np 0 smiles 0 ROMol 11 dtype: int64
يظهر الخطأ "التكافؤ الصريح للذرة # 1 N ، 4 ، أكبر من المسموح به" نظرًا لوجود جزيء (أيون ، إلخ) بتكافؤ غير طبيعي ("4 ذرات في N"). القيمة أعلى من المسموح به القيمة. ")لم يتم إرجاع أي منها إلى ROMol لمثل هذا الجزيء ، وكان هناك 11 من هذه الابتسامات هنا.
يمكنك التعامل معهم واحدًا تلو الآخر ، ولكن إذا كان الرقم صغيرًا ، فمن السهل إزالته في الوقت الحالي.لذلك ، استخدم isnull (). Sum () للتحقق من القيم المفقودة في عمود ROMol ، وإزالة تلك الصفوف إن وجدت.
المرجع: استكشاف أخطاء قراءة البيانات المركبة وإصلاحها
# ROMolが作成できなかったものを確認
print(df[df.ROMol.isnull()])
# 欠損行の除去
df = df.dropna() 
إذا رأيت "تحذير: عدم إزالة ذرات الهيدروجين بدون جيران" ، فربما يرجع ذلك إلى احتواء البيانات على أملاح. يحفظ RDKit H افتراضيًا ، لذلك إذا كان هناك H (مثل الملح) غير مرتبط بالجيران ، فلا يمكن إزالة H وتحذيرك.
إنشاء واصف جزيئي
تُعد وظيفة الخريطة مفيدة لتطبيق دالة على كائن في كل صف في إطار البيانات.
نظرًا لأن أسماء الواصفات والوظائف مدرجة في "Descriptors.descList" لـ RDKit ، فقد استغرق الأمر بعض الوقت ، لكنني تمكنت من حساب الكل مرة واحدة باستخدام دالة for و map وإعادتها إلى إطار البيانات.
for i,j in Descriptors.descList:
df[i] = df.ROMol.map(j)
df.shape
# (2039, 205)
df.head()
تمت إضافة واصفات لـ 201 عمودًا.
عند تطبيق المتغير الذي تم الحصول عليه على scikit-Learn أو إطار عمل التعلم العميق ، قد تحصل على الخطأ "خطأ في القيمة: يحتوي الإدخال على NaN أو ما لا نهاية أو قيمة كبيرة جدًا بالنسبة لـ dtype ('float64')". كان جيدًا إذا فعلت ما يلي .
for i,j in Descriptors.descList:
df[i] = df['ROMol'].map(j)
df['Ipc'] = [Descriptors.Ipc(mol, avg=True) for mol in df['ROMol']] يبدو أن السبب هو أن جزءًا من قيمة "IPC" للواصف يتم إنشاؤه بحجم لا نهائي.
المرجع: # 12 ماذا تفعل إذا كانت قيمة IPC لوصف RDKit 2D كبيرة جدًا
المرجع: قائمة الواصفات الجزيئية
إنشاء بصمة
يمكن حسابها بسرعة باستخدام وظيفة التطبيق ، ولكن يبدو أن قائمة بصمات الأصابع مخزنة في عمود واحد.
نظرًا لأنه يتم حفظ بصمة الإصبع بتنسيق كائن Explicit BitVect ، فقد استغرق الأمر بعض الوقت لتخزين كل قيمة في عمود واحد.
# 下準備
df = pd.read_csv("BBBP.csv")
PandasTools.AddMoleculeColumnToFrame(df,'smiles')
df = df.dropna()
# 1列にfingerprintのリストを追加する場合
df['FP'] = df.apply(lambda x: AllChem.GetMorganFingerprintAsBitVect(x.ROMol, 2, 1024), axis=1)
# fingerprintの各値を各列に格納する場合
# 個別に01をデータフレームに格納する
FP = [AllChem.GetMorganFingerprintAsBitVect(mol, 2, 1024) for mol in df.ROMol]
df_FP = pd.DataFrame(np.array(FP))
# フィンガープリントをもとのデータフレームに結合
df_FP.index = df.index
df = pd.concat([df, df_FP], axis=1)

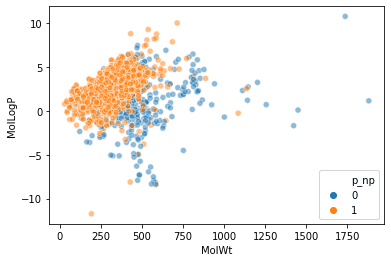









نقاش
قائمة التعليقات
لا توجد أي تعليقات حتى الآن