化学データセットの可視化と探索的データ解析【Python, RDKit】
機械学習/QSARモデル作成の前段階として、例えば化学構造と活性のリストからなる化合物データについて、その中身を俯瞰的に解析することはモデルの選択や理解のために重要です。
今回は、目的変数と関連のある特徴を見出すためのデータサイエンス手法として、化合物データセットの探索的データ解析を行ってみる。
探索的データ解析とは
探索的データ解析(Exploratory data analysis)は、データの可視化や相関解析など通して主な特徴を要約する、 データ分析の初期段階で重要なアプローチです。データの中からもともと想定していたパターンが認められるかを検証したり、何かしらのパターンがあるかを探したりするために行われます。
1970年代の統計学者Tukeyの書籍「Exploratory data analysis」は今や約2万件引用されているほど、データ分析において重要なプロセスとして定着している (Tukey , John W. Exploratory data analysis. Vol. 2. 1977. )。
データの準備
サンプルデータにはMoleculeNetのBBBPを利用。 blood-brain barrier penetration(化合物の血液脳関門透過性)について「透過性あり(penetration)」を1、「透過性なし(non-penetration)」を0でまとめたデータになります。
参考文献:https://pubs.acs.org/doi/10.1021/ci300124c
データの下準備
import numpy as np
import pandas as pd
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, PandasTools, Descriptors
pd.set_option('display.max_columns',250)
print('rdkit version: ',rdBase.rdkitVersion) #rdkit version: 2019.03.4
bbbp = pd.read_csv('./BBBP.csv',index_col=0)
# smilesからmolオブジェクトを作成し、DataFrameに格納
PandasTools.AddMoleculeColumnToFrame(bbbp,'smiles')
# molオブジェクトを作れなかった行の削除
bbbp = bbbp.dropna()
bbbp.info()<class 'pandas.core.frame.DataFrame'> Int64Index: 2039 entries, 1 to 2053 Data columns (total 4 columns): name 2039 non-null object p_np 2039 non-null int64 smiles 2039 non-null object ROMol 2039 non-null object dtypes: int64(1), object(3) memory usage: 79.6+ KB
データセットの俯瞰
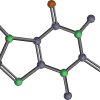
まずはどんな化学構造が含まれているのか一覧で確認します。
# 化学構造を一覧で表示
PandasTools.FrameToGridImage(bbbp[:18], column='ROMol', legendsCol='name', molsPerRow=6, subImgSize=(150,150))
# ターゲットp-npの分布を確認
bbbp.p_np.value_counts()1 1560 0 479 Name: p_np, dtype: int64
化合物記述子から目的変数との相関を探る
目視だけでは概要を捉えきれないので、記述子を作成・集計してデータセット俯瞰してみる。
化合物記述子については以下にまとめています。
# 記述子を生成する
for i,j in Descriptors.descList:
bbbp[i] = bbbp.ROMol.map(j)
# 要約統計量の表示
bbbp.describe()
# データセット全体における構造記述子の分布
import matplotlib.pyplot as plt
import math
list = ["p_np", "RingCount", "NumAromaticRings", "NumAromaticCarbocycles", "NumAromaticHeterocycles", "NumSaturatedCarbocycles", "NumSaturatedHeterocycles", "NumSaturatedRings"]
fig = plt.figure(figsize=(15,7))
for i, c in enumerate(list):
ax = fig.add_subplot(
math.ceil(len(list) / 4), 4, i + 1)
# plot the continent on these axes
sns.countplot(x=c, data=bbbp, ax=ax)
ax.set_title(c)
fig.tight_layout()
plt.show()
# targetによる構造記述子の分布の違いを可視化
list = ["RingCount", "NumAromaticRings", "NumAromaticCarbocycles", "NumAromaticHeterocycles", "NumSaturatedCarbocycles", "NumSaturatedHeterocycles", "NumSaturatedRings"]
fig = plt.figure(figsize=(15,7))
for i, c in enumerate(list):
ax = fig.add_subplot(
math.ceil(len(list) / 4), 4, i + 1)
# plot the continent on these axes
sns.countplot(x=c, data=bbbp, ax=ax, hue="p_np")
ax.set_title(c)
fig.tight_layout()
plt.show()
# logPと分子量でplotしてみる
import seaborn as sns
sns.scatterplot(x="MolWt", y="MolLogP", data=bbbp,
hue="p_np",
alpha=0.5)
血液脳関門透過性ということなので重要な因子はほぼ既知でしたが、疎水性(logP)と分子量(MolWt)が膜透過性に重要であるという関係性が伺えます。
一般的な探索的データ解析における手法として以下などが参考になりました( 探索的データ解析における正しい可視化手法の選び方と描き方 )。記述子の集計に適用するのもよさそう。









ディスカッション
コメント一覧
まだ、コメントがありません