SMILES से आणविक विवरणकों और उंगलियों के निशान की गणना करें और उन्हें एक डेटा फ्रेम [पायथन, आरडीकिट] में संग्रहीत करें
यह RDKit के साथ यौगिक डेटासेट SMILES से आणविक विवरणक और उंगलियों के निशान युक्त डेटा फ्रेम बनाने की एक विधि है। मैंने अपना खुद का QSAR/मशीन लर्निंग मॉडल बनाने की कोशिश की, लेकिन मैं आणविक विवरणक और उंगलियों के निशान बनाने में काफी हद तक लड़खड़ा गया, इसलिए मैं नीचे संक्षेप में बताऊंगा।
डेटा फ्रेम में स्टोर करने का महत्व
यदि यह केवल मशीन लर्निंग के लिए है, तो आप इसे सूची प्रकार के रूप में बना सकते हैं, लेकिन इसे डेटा फ़्रेम बनाने से निम्न कार्य करना आसान हो जाता है।
- निर्मित विवरणकों/फिंगरप्रिंट्स के आधार पर यौगिक डेटासेट का विहंगम दृश्य
- डेटा प्रीप्रोसेसिंग जैसे मिसिंग वैल्यू हैंडलिंग और डायमेंशनलिटी रिडक्शन
इसके अलावा, RDKit पहले डिस्क्रिप्टर की गणना करने के लिए SMILES को मोल ऑब्जेक्ट में परिवर्तित करता है, लेकिन डेटा फ़्रेम को संभालना आसान होता है, भले ही ऐसी चीज़ें हों जिन्हें अच्छी तरह से परिवर्तित नहीं किया जा सकता है।
कोशिश करके देखो
तैयारी
नमूना डेटा के लिए, हम मॉलिक्यूलनेट के बीबीबीपी (ब्लड-ब्रेन बैरियर पेनिट्रेशन डेटा सेट) SMIELS का उपयोग करेंगे।
RDKit mol ऑब्जेक्ट्स को ROMol नामक कॉलम में स्टोर किया जाता है, इसलिए इसके आधार पर एक डिस्क्रिप्टर बनाएं।
import numpy as np
import pandas as pd
from rdkit import rdBase, Chem
from rdkit.Chem import AllChem, PandasTools, Descriptors
from rdkit.Chem.Draw import IPythonConsole
print('rdkit version: ',rdBase.rdkitVersion) # rdkit version: 2019.03.4
# 下準備
# データセットの読み込み
df = pd.read_csv("BBBP.csv")
# dfのSMILES列を参照してMolオブジェクト列をデータフレームに加える
PandasTools.AddMoleculeColumnToFrame(df,'smiles')
# Molオブジェクトが作成できたか確認
print(df.shape)
print(df.isnull().sum()) (2050, 4) अंक 0 नाम 0 p_np 0 मुस्कान 0 रोमोल 11 dtype: int64
त्रुटि "परमाणु # 1 एन, 4 के लिए स्पष्ट वैलेंस, अनुमति से अधिक है" दिखाई देती है, लेकिन यह असामान्य वैलेंस ("एटम # 4 एन, 11, अनुमति से अधिक है) के साथ एक अणु (जैसे आयन) के कारण होता है "). मूल्य स्वीकार्य मूल्य से अधिक है")।इस तरह के एक अणु के लिए रोमोल में कोई भी नहीं लौटाया गया था, और यहां ऐसी XNUMX मुस्कानें थीं।
आप उनसे एक-एक करके निपट सकते हैं, लेकिन यदि संख्या कम है, तो उन्हें कुछ समय के लिए हटाना जल्दी होगा।इसलिए, isnull().sum() का उपयोग यह जांचने के लिए करें कि क्या ROMol कॉलम में कोई गुम मान है, और यदि है, तो पंक्ति को हटा दें।
संदर्भ: मिश्रित डेटा लोड करने में समस्या निवारण
# ROMolが作成できなかったものを確認
print(df[df.ROMol.isnull()])
# 欠損行の除去
df = df.dropna() 
यदि आप "चेतावनी: पड़ोसियों के बिना हाइड्रोजन परमाणु को नहीं हटा रहे हैं" देखते हैं, तो यह संभवतः इसलिए है क्योंकि डेटा में नमक है। डिफ़ॉल्ट रूप से, RDKit H को हटाकर डेटा सहेजता है, इसलिए यदि कोई H (जैसे नमक) है जो पड़ोसी के साथ बंधता नहीं है, तो ऐसे H को हटाया नहीं जा सकता है और एक चेतावनी जारी की जाती है।
आणविक विवरणकों का निर्माण
डेटाफ़्रेम में प्रत्येक पंक्ति में फ़ंक्शन लागू करने के लिए मैप फ़ंक्शन उपयोगी है।
चूंकि डिस्क्रिप्टर के नाम और फ़ंक्शंस RDKit के "Descriptors.descList" में सूचीबद्ध हैं, इसमें कुछ समय लगता है, लेकिन मैं फ़ंक्शन और मैप फ़ंक्शन के लिए बैच गणना करने में सक्षम था और उन्हें डेटा फ़्रेम में लौटाता था।
for i,j in Descriptors.descList:
df[i] = df.ROMol.map(j)
df.shape
# (2039, 205)
df.head()
201 कॉलम के लिए विवरणक जोड़े गए।
प्राप्त चर को स्किकिट-लर्न या डीप लर्निंग फ्रेमवर्क में लागू करते समय, आपको एक त्रुटि मिल सकती है "ValueError: इनपुट में NaN, अनंत या एक मान dtype ('फ्लोट 64') के लिए बहुत बड़ा है", लेकिन उस स्थिति में यह ठीक था अगर मैंने किया:
for i,j in Descriptors.descList:
df[i] = df['ROMol'].map(j)
df['Ipc'] = [Descriptors.Ipc(mol, avg=True) for mol in df['ROMol']] ऐसा लगता है कि कारण यह है कि वर्णनकर्ता के एक हिस्से के "आईपीसी" मूल्य में एक असीम रूप से बड़ा मूल्य बनाया गया है।
संदर्भ: #12 जब RDKit 2D डिस्क्रिप्टर में IPC मान बहुत बड़ा हो तो क्या करें
संदर्भ: आण्विक वर्णनकर्ता सूची
एक फिंगरप्रिंट बनाएं
इसे लागू फ़ंक्शन का उपयोग करके जल्दी से गणना की जा सकती है, लेकिन ऐसा लगता है कि फ़िंगरप्रिंट की सूची एक कॉलम में संग्रहीत है।
फ़िंगरप्रिंट्स ExplicitBitVect ऑब्जेक्ट स्वरूप में संग्रहीत किए जाते हैं, इसलिए प्रत्येक मान को एक पंक्ति में संग्रहीत करना थोड़ा परेशानी भरा था।
# 下準備
df = pd.read_csv("BBBP.csv")
PandasTools.AddMoleculeColumnToFrame(df,'smiles')
df = df.dropna()
# 1列にfingerprintのリストを追加する場合
df['FP'] = df.apply(lambda x: AllChem.GetMorganFingerprintAsBitVect(x.ROMol, 2, 1024), axis=1)
# fingerprintの各値を各列に格納する場合
# 個別に01をデータフレームに格納する
FP = [AllChem.GetMorganFingerprintAsBitVect(mol, 2, 1024) for mol in df.ROMol]
df_FP = pd.DataFrame(np.array(FP))
# フィンガープリントをもとのデータフレームに結合
df_FP.index = df.index
df = pd.concat([df, df_FP], axis=1)

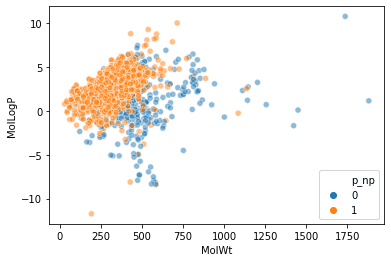








विचार-विमर्श
टिप्पणियों की सूची
अभी तक कोई टिप्पणी नहीं है