Визуализируйте составное пространство за счет уменьшения размерности отпечатка пальца
Давайте визуализируем составное пространство (химическое пространство) на основе химической структуры, используя набор данных соединения, открытый для публики.
Мотивация
Визуализация составного пространства
-Есть ли в наборе данных какие-то уникальные соединения?
・ Какого рода соединение содержится и в каком количестве
Это поможет вам понять. При создании прогнозных моделей для QSAR и машинного обучения вы можете примерно определить, есть ли у ваших данных экстраполяция.
метод
Расчет отпечатка пальца
Создав отпечаток пальца для каждого соединения и уменьшив размер, можно построить график на плоскости. Согласно принципу схожести свойств Джонсона и Маджоры «Подобные соединения обладают схожими свойствами», соединения с подобными структурами и свойствами должны располагаться близко друг к другу на плоскости.
В качестве отпечатка пальца, используемого для визуализации, попробуйте отпечаток пальца Моргана и отпечаток RDkit.
Ссылка: отпечаток пальца доступен в RDkit
Метод уменьшения размерности
Для уменьшения размерности попробуйте использовать анализ главных компонентов (PCA) и UMAP. Чаще всего используется PCA, но он сжимается в пространство более низкой размерности на основе линейности данных, поэтому он может не подходить для данных 0-1, таких как отпечатки пальцев.
С другой стороны, UMAP - это один из методов уменьшения размерности, учитывающий нелинейные компоненты.Снижение размерности, подобное стандартному t-SNE того же метода, может быть выполнено в несколько раз быстрее, поэтому его можно использовать для больших наборов данных.
Используйте метод kmeans и спектральную кластеризацию для кластеризации.
Метод kmeans объединяется в кластеры путем повторения ряда операций: (XNUMX) случайная установка кластеров, (XNUMX) добавление ближайших точек данных для обновления положения центра тяжести и (XNUMX) повторное нахождение центра тяжести с обновленными данными рядом с центр тяжести. Известно, что нелинейные данные, такие как луна и швейцарский рулет, не могут быть хорошо классифицированы, но Spectral Clustering, похоже, может обрабатывать такие данные. Даже шпаргалка scikit learn - вариант, если метод kmeans не работает.
Я хотел бы объединить каждый из них с «методом PCA x kmeans», который предполагает нормальное распределение и линейные данные, и «UMAP x Spectral Clustering», который поддерживает нелинейные данные.
Кстати, существует еще метод кластеризации соединений на основе коэффициента танимото (сходство между двумя соединениями выражается от 2 до 0. Структура такая же, как у 1), который часто используется как показатель подобия соединений, но в этот раз попробовал. не ...
Ссылка: уменьшение размерности и методы построения 2D-графиков для данных большой размерности
Спектральная кластеризация (кластерный анализ)
Визуализируем составное пространство
Используйте липофильность Molecule net для набора данных.Приблизительно 4200 соединений содержат соответствующие SMILES и экспериментальный logP (индекс гидрофобности: коэффициент распределения октанол / вода).
Смотрите также:Список составных наборов данных
# пакет import import pandas as pd import numpy as np from sklearn.cluster import KMeans from sklearn.decomposition import PCA from sklearn.cluster import SpectralClustering import umap from rdkit import Chem from rdkit.Chem import AllChem import matplotlib.pyplot as plt% matplotlib inline # читать набор данных df = pd.read_csv ('Lipophilicity.csv') print (df.info ()) df.head (5)
RangeIndex: 4200 записей, от 0 до 4199
Столбцы данных (всего 3 столбца):
CMPD_CHEMBLID 4200 ненулевой объект
exp 4200 ненулевое значение float64
улыбается 4200 ненулевой объект
dtypes: float64 (1), объект (2) использование памяти: 98.5+ КБ Нет

#Morgan and RDkit Get Fingerprint mols = [Chem.MolFromSmiles (x) для x в df.smiles] morgan_fps = [AllChem.GetMorganFingerprintAsBitVect (x, 2, 1024) для x в mols] rdkit_fps = [Chem.RDKFpSprintize (x, = 1024) для x в mols] # сохранить отпечаток пальца в DF df_morgan_fps = pd.DataFrame (np.array (morgan_fps)) df_rdkit_fps = pd.DataFrame (np.array (rdkit_fps))
По отпечатку пальца Моргана подсчитываются подструктуры в пределах заданного радиуса от атома. Алгоритм похож на отпечаток ECFP (Extended Connectivity Fingerprint), где радиус 2 Моргана соответствует ECFP4.Здесь мы вычисляем отпечаток с радиусом = 2 бит.
Отпечаток RDkit считает частичные структуры на основе длины связи, а не радиуса от атома.Это похоже на отпечаток Daylight.По умолчанию минимальная длина пути: 1 соединение - максимальная длина пути: рассматриваются 7 соединений.
Полученный отпечаток пальцаПоскольку последующая обработка не может быть выполнена, как с таким объектом, исправьте ее до фрейма данных.
# kmeans clustering kmeans = KMeans (n_clusters = 8, n_jobs = -1) kmeans.fit (df_morgan_fps) # Уменьшение размеров с помощью PCA pca = PCA (n_components = 2) decomp = pca.fit_transform (df_morgan_fps) x = decomp [:, 0 ] y = decomp [:, 1] #Visualization plt.figure (figsize = (15,5)) # Цветные кластеры, полученные с помощью kmeans plt.subplot (1,2,1) plt.scatter (x, y), c = kmeans.labels_, alpha = 0.7) plt.title ("PCA: morgan_fps, cluster") plt.colorbar () # цветовая кодировка с помощью logP plt.subplot (1,2,2) plt.scatter (x, y, c = df.exp, alpha = 0.7, cmap = 'spring') plt.title ("PCA: morgan_fps, logP") plt.colorbar ()
Результат визуализации составного пространства с помощью отпечатка RDkit.

Точно так же измените часть df_morgan_fps на df_rdkit_fps и визуализируйте ее с помощью отпечатка RDkit.Как-то впечатление, что отпечаток RDkit проецируется красивее ↓

# Отпечатки RDkit, визуализированные с помощью "UMAP x Spectral Clustering" sc = SpectralClustering (n_clusters = 50, affinity = 'near_neighbors', n_jobs = -1) sc.fit (df_rdkit_fps) embedding = umap.UMAP (n_neighbors = 50, n_components = 2, min_dist = 0.5) .fit_transform (df_rdkit_fps) x = embedding [:, 0] y = embedding [:, 1] fig = plt.figure (figsize = (15,5)) plt.subplot (1,2,1)) plt.scatter (x, y, c = sc.labels_, alpha = 0.7) plt.title ("rdkit_fps, SpectralCluster") plt.colorbar () plt.subplot (1,2,2) plt.scatter (x, y) , c = df.exp, alpha = 0.7, cmap = 'spring') plt.title ("rdkit_fps, logP") plt.colorbar ()

Мне кажется, что «UMAP x Spectral Clustering» более плотно заполнен подобными соединениями, чем «PCA x kmeans», но любой из них подходит.
Что касается logP, связь между положением на плоскости и значением физического свойства очень тонкая.Автоэнкодер или контролируемое обучение, кажется, лучше подходят для построения графиков в двумерном пространстве в лучшей корреляции с физическими свойствами и значениями активности соединения, поэтому я хотел бы вскоре попробовать его.




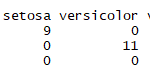





обсуждение
Список комментариев
Пока нет комментариев