Pruebe un modelo QSAR simple con Chainer [Predicción de la permeabilidad de los compuestos en la barrera hematoencefálica]
QSAR (relación cuantitativa estructura-actividad) es una correlación estadística entre la estructura de una sustancia química y su actividad fisiológica (toxicidad, capacidad de unirse a enzimas, actividad como fármaco, etc.).El rendimiento de los compuestos se puede predecir a partir de correlaciones basadas en amplios conjuntos de datos experimentales de sustancias químicas.
Esta vez, crearemos un modelo QSAR simple que "predice la permeabilidad de la barrera hematoencefálica de los compuestos" utilizando Chainer, un marco de aprendizaje profundo fabricado en Japón, y verificaremos su rendimiento frente al conjunto de pruebas.
Destino y datos de predicción
Para los datos, se utiliza BBBP de Molecule Net.Consulte a continuación para obtener una vista panorámica de los datos.
Con respecto a la permeabilidad de la barrera hematoencefálica del compuesto, "penetración" es XNUMX y "no penetración" es XNUMX.
Creando un modelo
环境
from rdkit import rdBase
import chainer
print('rdkit version: ',rdBase.rdkitVersion)
chainer.print_runtime_info()rdkit versión: 2019.03.4 Plataforma: Linux-5.0.0-37-generic-x86_64-with-debian-buster-sid Chainer: 6.2.0 NumPy: 1.17.4 CuPy: CuPy Versión: 6.2.0 CUDA Raíz: / usr / local / cuda CUDA Versión de compilación: 10010 CUDA Versión del controlador: 10010 CUDA Versión en tiempo de ejecución: 10010 cuDNN Versión de compilación: 7500 cuDNN Versión: 7605 NCCL Versión de compilación: 2402 NCCL Versión en tiempo de ejecución: 2402 iDeep: No disponible
Puede comprobar la versión de Chainer, Numpy, Cupy utilizada en chainer.print_runtime_info ().
Construcción del modelo
import numpy as np
import pandas as pd
from rdkit import Chem
from rdkit.Chem import Draw, PandasTools, Descriptors
# データの読み込み
df = pd.read_csv('BBBP.csv',index_col=0)
# smilesからmolファイルを生成し、データフレーム中に加える
PandasTools.AddMoleculeColumnToFrame(df, smilesCol = 'smiles')
# molができなかった行を削除する
df = df.dropna()
# molファイルから化合物記述子を算出する
for i,j in Descriptors.descList:
df[i] = df['ROMol'].map(j)
df['Ipc'] = [Descriptors.Ipc(mol, avg=True) for mol in df['ROMol']]
# chainer用にデータ型を変換
x = df.iloc[:,4:].values.astype('float32')
y = df['p_np'].values.astype('int32')
indices = np.array(range(x.shape[0])) # train_test_split後も列番号を保持しておく
# train, test, valに分割
from sklearn.model_selection import train_test_split
x_train, x_test, y_train, y_test, indices_train, indices_test = train_test_split(x, y, indices, test_size=0.05, random_state=123)
# 説明変数の標準化
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
scaler.fit(x_train)
x_train= scaler.transform(x_train)
x_test = scaler.transform(x_test)
print(type(x_train), x_train.shape, type(y_train), y_train.shape)
print(type(x_test), x_test.shape, type(y_test), y_test.shape)(1937, 200) (1937,) (102, 200) (102,)
Consulte aquí para crear descriptores a partir de objetos mol.
Calcule el descriptor molecular y la huella digital de SMILES y guárdelo en el marco de datos [Python, RDKit]
Los tutoriales oficiales sobre cómo usar Chainer son muy extensos.
Introducción al tutorial de Deep Learning Chainer
# 説明変数と目的変数のセットで使えるように変換する
from chainer.datasets import TupleDataset
train = TupleDataset(x_train, y_train)
test = TupleDataset(x_test, y_test)
# イテレータの準備
from chainer.iterators import SerialIterator
train_iter = SerialIterator(train, batch_size=64, repeat=True, shuffle=True)
test_iter = SerialIterator(test, batch_size=64, shuffle=False, repeat=False)
# ニューラルネットワークの作成
# 3層のmulti layer perceptron(MLP)
import chainer.links as L
import chainer.functions as F
from chainer import Chain
from chainer import optimizers, training
from chainer.training import extensions
class MLP(chainer.Chain):
def __init__(self):
super().__init__()
with self.init_scope():
self.fc1 = L.Linear(None, 100)
self.fc2 = L.Linear(None, 20)
self.fc3 = L.Linear(None, 2)
def forward(self, x):
h = F.relu(self.fc1(x))
h = F.relu(self.fc2(h))
h = self.fc3(h)
return h
# ネットワークをClassifierでラップしする
# (目的関数(デフォルトはsoftmax交差エントロピー)の計算し、損失を返す)
predictor = MLP()
net = L.Classifier(predictor)
# 最適化手法を選択して、オプティマイザを作成する
optimizer = optimizers.MomentumSGD(lr=0.1).setup(net)
# アップデータにイテレータとオプティマイザを渡す
updater = training.StandardUpdater(train_iter, optimizer, device=-1)
trainer = training.Trainer(updater, (50, 'epoch'), out='/results/')from chainer.training import extensions
trainer.extend(extensions.LogReport(trigger=(5, 'epoch'), log_name='log'))
trainer.extend(extensions.snapshot(filename='snapshot_epoch-{.updater.epoch}'))
trainer.extend(extensions.dump_graph('main/loss'))
trainer.extend(extensions.Evaluator(test_iter, net, device=-1), name='val')
trainer.extend(extensions.PrintReport(['epoch', 'iteration', 'main/loss', 'main/accuracy', 'val/main/loss', 'val/main/accuracy', 'fc1/W/data/mean', 'elapsed_time']))
trainer.extend(extensions.PlotReport(['fc1/W/grad/mean'], x_key='epoch', file_name='mean.png'))
trainer.extend(extensions.PlotReport(['main/loss', 'val/main/loss'], x_key='epoch', file_name='loss.png'))
trainer.extend(extensions.PlotReport(['main/accuracy', 'val/main/accuracy'], x_key='epoch', file_name='accuracy.png'))
trainer.extend(extensions.ParameterStatistics(net.predictor.fc1, {'mean': np.mean}, report_grads=True))
trainer.run()from IPython.display import Image, display
display(Image(filename='results/accuracy.png'))
Parece que la precisión es razonable, pero la precisión del conjunto de prueba no ha mejorado mucho incluso a medida que avanza el aprendizaje ...
inferencia
# 学習したモデルで推論してみる
with chainer.using_config('train', False), chainer.using_config('enable_backprop', False):
y_pred = predictor(x_test)
# 推論結果の確認
print('accuracy', F.accuracy(y_pred, y_test)) # accuracy variable(0.88235295)
from sklearn.metrics import confusion_matrix
confusion_matrix(y_test, y_pred.data.argmax(axis=1))matriz ([[21, 7], [5, 69]])
la precisión es un valor como puede ver en la figura.Al evaluar la precisión de la clasificación por la matriz de confusión, existen falsos positivos y falsos negativos, pero parece que la precisión no la gana la clasificación que está sesgada a un lado.
# 一部予測結果を見てみる
for i in range(int(len(y_pred)/10)):
print('No.', indices_test[i])
print('label:', y_test[i])
print('pred :', np.argmax(y_pred[i].array))
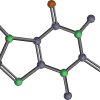
img = Draw.MolToImage(df.ROMol[indices_test[i]])
display(img)
La imagen es parte del resultado, pero la respuesta correcta es que el ojo humano puede discernirla.Parece divertido escudriñar qué cometiste un error.
![Visualización de conjuntos de datos químicos y análisis de datos exploratorios [Python, RDKit]](https://insilico-notebook.com/wp-content/uploads/2020/02/image-4-150x150.png)









discusión
Lista de comentarios
Aún no hay comentarios