Visualize o espaço composto por redução da dimensionalidade da Fingeprint
Vamos visualizar o espaço composto (espaço químico) com base na estrutura química usando o conjunto de dados composto que está aberto ao público.
Motivação
Visualização do espaço composto
-Há algum composto exclusivo no conjunto de dados?
・ Que tipo de composto está contido e quanto
Isso o ajudará a entender. Ao criar modelos preditivos para QSAR e aprendizado de máquina, você pode determinar aproximadamente se seus dados têm extrapolações.
方法
Cálculo de impressão digital
Ao gerar uma impressão digital para cada composto e reduzir a dimensão, é possível plotar em um plano. De acordo com o princípio de propriedades semelhantes de Johnson e Maggiora, "Compostos semelhantes têm propriedades semelhantes", compostos com estruturas e propriedades semelhantes devem ser distribuídos próximos uns dos outros em um plano.
Para a impressão digital usada para visualização, experimente a impressão digital Morgan e a impressão digital RDkit.
Referência: impressão digital disponível no RDkit
Método de redução de dimensionalidade
Para redução de dimensionalidade, tente usar Análise de Componentes Principais (PCA) e UMAP. O PCA é mais comumente usado, mas é compactado em um espaço dimensional inferior com base na linearidade dos dados, portanto, pode não ser adequado para dados 0-1, como impressões digitais.
Por outro lado, o UMAP é um dos métodos de redução de dimensão considerando componentes não lineares.A redução da dimensionalidade, como o t-SNE padrão do mesmo método, pode ser concluída várias vezes mais rápido, portanto, pode ser usada para grandes conjuntos de dados.
Use o método kmeans e Spectral Clustering para agrupamento.
O método kmeans se agrupa repetindo uma série de operações: (XNUMX) instalação aleatória de clusters, (XNUMX) adição de pontos de dados próximos para atualizar a posição do centro de gravidade e (XNUMX) encontrar o centro de gravidade novamente com os dados atualizados próximos o centro de gravidade. Sabe-se que dados não lineares como Lua e Rolinho Suíço não podem ser bem classificados, mas o Spectral Clustering parece ser capaz de lidar com tais dados. Mesmo a folha de dicas do scikit learn é uma opção se o método kmeans não funcionar.
Gostaria de combinar cada um deles com o "método PCA x kmeans" que assume distribuição normal e dados lineares e o "UMAP x Spectral Clustering" que suporta dados não lineares.
A propósito, há também um método de agrupamento de compostos com base no coeficiente de tanimoto (a similaridade entre dois compostos é expressa de 2 a 0. A mesma estrutura de 1), que é frequentemente usado como um índice de similaridade de compostos, mas desta vez eu tentei. Não é ...
Referência: Redução de dimensionalidade e técnicas de plotagem 2D para dados de alta dimensão
Clustering espectral (análise de cluster)
Vamos visualizar o espaço composto
Use a lipofilicidade da rede Molecule para o conjunto de dados.Aproximadamente 4200 compostos contêm seus respectivos SMILES e logP experimental (índice de hidrofobicidade: coeficiente de partição octanol / água).
Referência:Lista de conjuntos de dados compostos
# package import import pandas as pd import numpy as np from sklearn.cluster import KMeans from sklearn.decomposition import PCA from sklearn.cluster import SpectralClustering import umap from rdkit import Chem from rdkit.Chem import AllChem import matplotlib.pyplot as plt% matplotlib inline # read dataset df = pd.read_csv ('Lipophilicity.csv') print (df.info ()) df.head (5)
RangeIndex: 4200 entradas, 0 a 4199
Colunas de dados (total de 3 colunas):
Objeto não nulo CMPD_CHEMBLID 4200
exp 4200 não nulo float64
sorri 4200 objeto não nulo
dtypes: float64 (1), objeto (2) uso de memória: 98.5+ KB Nenhum

#Morgan e RDkit Obter mols de impressão digital = [Chem.MolFromSmiles (x) para x em df.smiles] morgan_fps = [AllChem.GetMorganFingerprintAsBitVect (x, 2, 1024) para x em mols] rdkit_fps = [Chem.RDKFingerprint (x, fpSize = 1024) para x em mols] #store impressão digital em DF df_morgan_fps = pd.DataFrame (np.array (morgan_fps)) df_rdkit_fps = pd.DataFrame (np.array (rdkit_fps))
A impressão digital Morgan conta subestruturas dentro de um raio definido de um átomo. Um algoritmo semelhante à impressão digital ECFP (Extended Connectivity Fingerprint), onde o raio de Morgan 2 corresponde a ECFP4.Aqui, calculamos a impressão digital de raio = 2 bits.
A impressão digital RDkit conta estruturas parciais com base no comprimento da ligação, em vez do raio do átomo.Isso é semelhante à impressão digital da luz do dia.Por padrão, comprimento mínimo do caminho: 1 junção - comprimento máximo do caminho: 7 junções são consideradas.
A impressão digital obtida éUma vez que o processamento subsequente não pode ser executado como é com um objeto como, corrija-o para um quadro de dados.
# kmeans clustering kmeans = KMeans (n_clusters = 8, n_jobs = -1) kmeans.fit (df_morgan_fps) # Redução da dimensão com PCA pca = PCA (n_components = 2) decomp = pca.fit_transform (df_morgan_fps) x = decomp [:, 0 ] y = decomp [:, 1] #Visualization plt.figure (figsize = (15,5)) # Clusters codificados por cores obtidos por kmeans plt.subplot (1,2,1) plt.scatter (x, y), c = kmeans.labels_, alpha = 0.7) plt.title ("PCA: morgan_fps, cluster") plt.colorbar () # codificado por cor com logP plt.subplot (1,2,2) plt.scatter (x, y, c = df.exp, alpha = 0.7, cmap = 'spring') plt.title ("PCA: morgan_fps, logP") plt.colorbar ()
O resultado da visualização do espaço composto com a impressão digital RDkit.

Da mesma forma, altere a parte df_morgan_fps para df_rdkit_fps e visualize-o com a impressão digital RDkit.De alguma forma, a impressão de que a impressão digital do RDkit é projetada de forma mais bonita ↓

# RDkit Impressões digitais visualizadas com "UMAP x Spectral Clustering" sc = SpectralClustering (n_clusters = 50, affinity = 'mais próximo_neighs', n_jobs = -1) sc.fit (df_rdkit_fps) embedding = umap.UMAP (n_neighbors = 50, n_components = 2, min_dist = 0.5) .fit_transform (df_rdkit_fps) x = incorporação [:, 0] y = incorporação [:, 1] fig = plt.figure (figsize = (15,5)) plt.subplot (1,2,1)) plt.scatter (x, y, c = sc.labels_, alpha = 0.7) plt.title ("rdkit_fps, SpectralCluster") plt.colorbar () plt.subplot (1,2,2) plt.scatter (x, y , c = df.exp, alpha = 0.7, cmap = 'spring') plt.title ("rdkit_fps, logP") plt.colorbar ()

Eu sinto que "UMAP x Spectral Clustering" é mais densamente embalado com compostos semelhantes do que "PCA x kmeans", mas qualquer um está bem.
Em relação ao logP, a relação entre a posição no plano e o valor da propriedade física é delicada.Autoencoder ou aprendizado supervisionado parece ser melhor para plotar em um espaço bidimensional em correlação com as propriedades físicas e valores de atividade de compostos, então eu gostaria de tentar em breve.




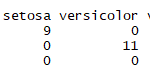





discussão
Lista de comentários
Ainda não há comentários