Visualizza lo spazio composto riducendo la dimensionalità di Fingeprint
Visualizziamo lo spazio composto (spazio chimico) in base alla struttura chimica utilizzando il set di dati composto che è aperto al pubblico.
Motivazione
Visualizzazione dello spazio composto
-Ci sono composti unici nel set di dati?
・ Che tipo di composto è contenuto e quanto
Ti aiuterà a capire. Quando si creano modelli predittivi per QSAR e machine learning, è possibile determinare approssimativamente se i dati hanno estrapolazioni.
方法
Calcolo delle impronte digitali
Generando un'impronta digitale per ogni composto e riducendo la dimensione, è possibile tracciare su un piano. Secondo il principio delle proprietà simili di Johnson e Maggiora, "Composti simili hanno proprietà simili", i composti con strutture e proprietà simili dovrebbero essere distribuiti uno vicino all'altro su un piano.
Per l'impronta digitale utilizzata per la visualizzazione, prova l'impronta digitale Morgan e l'impronta digitale RDkit.
Riferimento: impronta digitale disponibile in RDkit
Metodo di riduzione della dimensionalità
Per la riduzione della dimensionalità, provare a utilizzare Principal Component Analysis (PCA) e UMAP. La PCA è più comunemente usata, ma si comprime in uno spazio dimensionale inferiore in base alla linearità dei dati, quindi potrebbe non essere adatta per dati 0-1 come le impronte digitali.
D'altra parte, UMAP è uno dei metodi di riduzione delle dimensioni considerando i componenti non lineari.La riduzione della dimensionalità come il t-SNE standard dello stesso metodo può essere completata molte volte più velocemente, quindi può essere utilizzata per set di dati di grandi dimensioni.
Usa il metodo kmeans e il clustering spettrale per il clustering.
Il metodo kmeans raggruppa i cluster ripetendo una serie di operazioni: (XNUMX) impostando casualmente i cluster, (XNUMX) aggiungendo punti dati vicini per aggiornare la posizione del centro di gravità e (XNUMX) ritrovando il centro di gravità con i dati aggiornati vicino il centro di gravità. È noto che dati non lineari come Moon e Swiss roll non possono essere classificati bene, ma il clustering spettrale sembra essere in grado di gestire tali dati. Anche lo scikit learn cheat sheet è un'opzione se il metodo kmeans non funziona.
Vorrei combinare ciascuno di essi con il "metodo PCA x kmeans" che presuppone distribuzione normale e dati lineari e "UMAP x Spectral Clustering" che supporta dati non lineari.
A proposito, esiste anche un metodo di raggruppamento dei composti basato sul coefficiente tanimoto (la somiglianza tra due composti è espressa da 2 a 0. La stessa struttura di 1), che viene spesso utilizzata come indice di somiglianza dei composti, ma questa volta l'ho provato. Non è ...
Riferimento: tecniche di riduzione della dimensionalità e plottaggio 2D per dati ad alta dimensionalità
Spectral Clustering (analisi dei cluster)
Visualizziamo lo spazio composto
Usa Lipophilicity di Molecule net per il set di dati.Circa 4200 composti contengono i rispettivi SMILES e logP sperimentale (indice di idrofobicità: coefficiente di ripartizione ottanolo / acqua).
Vedi anche:Elenco dei set di dati composti
# package import import pandas come pd import numpy as np da sklearn.cluster import KMeans da sklearn.decomposition import PCA da sklearn.cluster import SpectralClustering import umap da rdkit import Chem da rdkit.Chem import AllChem import matplotlib.pyplot as plt% matplotlib inline # read dataset df = pd.read_csv ('Lipophilicity.csv') print (df.info ()) df.head (5)
RangeIndex: 4200 voci, da 0 a 4199
Colonne dati (totale 3 colonne):
CMPD_CHEMBLID 4200 oggetto non nullo
exp 4200 non nullo float64
sorride 4200 oggetto non nullo
dtypes: float64 (1), utilizzo della memoria object (2): 98.5+ KB Nessuno

#Morgan e RDkit Get Fingerprint mols = [Chem.MolFromSmiles (x) for x in df.smiles] morgan_fps = [AllChem.GetMorganFingerprintAsBitVect (x, 2, 1024) for x in mols] rdkit_fps = [Chem.RDKFingerprint (x, fpSize = 1024) for x in mols] # Memorizza l'impronta digitale in DF df_morgan_fps = pd.DataFrame (np.array (morgan_fps)) df_rdkit_fps = pd.DataFrame (np.array (rdkit_fps))
L'impronta digitale Morgan conta le sottostrutture entro un raggio impostato da un atomo. Un algoritmo simile all'impronta digitale ECFP (Extended Connectivity Fingerprint), dove il raggio 2 di Morgan corrisponde a ECFP4.Qui, calcoliamo l'impronta digitale del raggio = 2 bit.
L'impronta digitale RDkit conta le strutture parziali in base alla lunghezza del legame piuttosto che al raggio dall'atomo.È simile all'impronta digitale Daylight.Per impostazione predefinita, lunghezza minima del percorso: 1 join - lunghezza massima del percorso: vengono considerati 7 join.
L'impronta digitale ottenuta èPoiché la successiva elaborazione non può essere eseguita così com'è con un oggetto simile, correggerla in un frame di dati.
# kmeans clustering kmeans = KMeans (n_clusters = 8, n_jobs = -1) kmeans.fit (df_morgan_fps) # Riduzione dimensione con PCA pca = PCA (n_components = 2) decomp = pca.fit_transform (df_morgan_fps) x = decomp [:, 0 ] y = decomp [:, 1] #Visualization plt.figure (figsize = (15,5)) # Cluster con codice colore ottenuti da kmeans plt.subplot (1,2,1) plt.scatter (x, y), c = kmeans.labels_, alpha = 0.7) plt.title ("PCA: morgan_fps, cluster") plt.colorbar () # codificato a colori con logP plt.subplot (1,2,2) plt.scatter (x, y, c = df.exp, alpha = 0.7, cmap = 'spring') plt.title ("PCA: morgan_fps, logP") plt.colorbar ()
Il risultato della visualizzazione dello spazio composto con l'impronta digitale RDkit.

Allo stesso modo, cambia la parte df_morgan_fps in df_rdkit_fps e visualizzala con l'impronta digitale RDkit.In qualche modo l'impressione che l'impronta digitale RDkit sia proiettata in modo più bello ↓

# RDkit Impronte digitali visualizzate con "UMAP x Spectral Clustering" sc = SpectralClustering (n_clusters = 50, affinity = 'near_neighbors', n_jobs = -1) sc.fit (df_rdkit_fps) embedding = umap.UMAP (n_neighbors = 50, n_components = 2, min_dist = 0.5) .fit_transform (df_rdkit_fps) x = embedding [:, 0] y = embedding [:, 1] fig = plt.figure (figsize = (15,5)) plt.subplot (1,2,1)) plt.scatter (x, y, c = sc.labels_, alpha = 0.7) plt.title ("rdkit_fps, SpectralCluster") plt.colorbar () plt.subplot (1,2,2) plt.scatter (x, y , c = df.exp, alpha = 0.7, cmap = 'spring') plt.title ("rdkit_fps, logP") plt.colorbar ()

Ritengo che "UMAP x Spectral Clustering" sia più densamente imballato con composti simili rispetto a "PCA x kmeans", ma uno dei due va bene.
Per quanto riguarda logP, il rapporto tra la posizione sul piano e il valore della proprietà fisica è delicato.L'autoencoder o l'apprendimento supervisionato sembra essere migliore per tracciare in uno spazio bidimensionale in correlazione con le proprietà fisiche e i valori di attività dei composti, quindi vorrei provarlo presto.




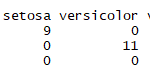





discussione
Elenco dei commenti
Non ci sono ancora commenti